Produits populaires
-

PMSF BioChemica
A0999 -

IPTG BioChemica
A1008 -

DTT BioChemica
A1101 -

NBT BioChemica
A1243 -

-

104 produits disponibles
- 2-Nitrophényl-ß-D-Galactopyranoside BioChemica A1272,0025 · 369-07-3
- 3,3',5,5'-Tétraméthylbenzidine BioChemicaA3840,0025 · 54827-17-7
- ABTS® BioChemica A1088,0010 · 30931-67-0
- ABTS® BioChemica A1088,0005 · 30931-67-0
- Acrylamide - Solution (30 %) - Mélange 29 : 1 pour biologie moléculaireA4983,0500 ·
- Acrylamide - Solution (30 %) - Mélange 37,5 : 1 pour la biologie moléculaireA3626,0500 ·
- Acrylamide - Solution (30 %) - Mix 37.5 : 1 pour la biologie moléculaireA3626,1000 ·
- Acrylamide - Solution (40 %) - Mélange 37,5 : 1 pour la biologie moléculaireA4989,0500 ·
- Acrylamide - Solution (40 %) - Mix 19 : 1 pour la biologie moléculaireA3658,1000 ·
- Acrylamide - Solution (40 %) - Mix 29 : 1 pour la biologie moléculaireA0385,1000 ·
- Acrylamide - Solution (40 %) - Mix 29 : 1 pour la biologie moléculaireA0385,0500 ·
- Acrylamide 2K Standard grade, pur A1089,0500 · 79-06-1
- Acrylamide 4K - Solution (30 %) - Mélange 29 : 1 A0951,1000 ·
- Acrylamide 4K - Solution (30 %) - Mélange 37.5 : 1 A1672,1000 ·
- Acrylamide 4K - Solution (40 %) - Mix 37.5 : 1 A1577,1000 ·
Biochimie des protéines
On trouve des protéines dans chaque cellule. Leur composition est cartographiée par le code génétique. Les gènes sont lus au cours de ce que l'on appelle l'expression, afin que les protéines soient disponibles et fonctionnelles dans l'être vivant.
Les protéines sont constituées d'acides aminés.
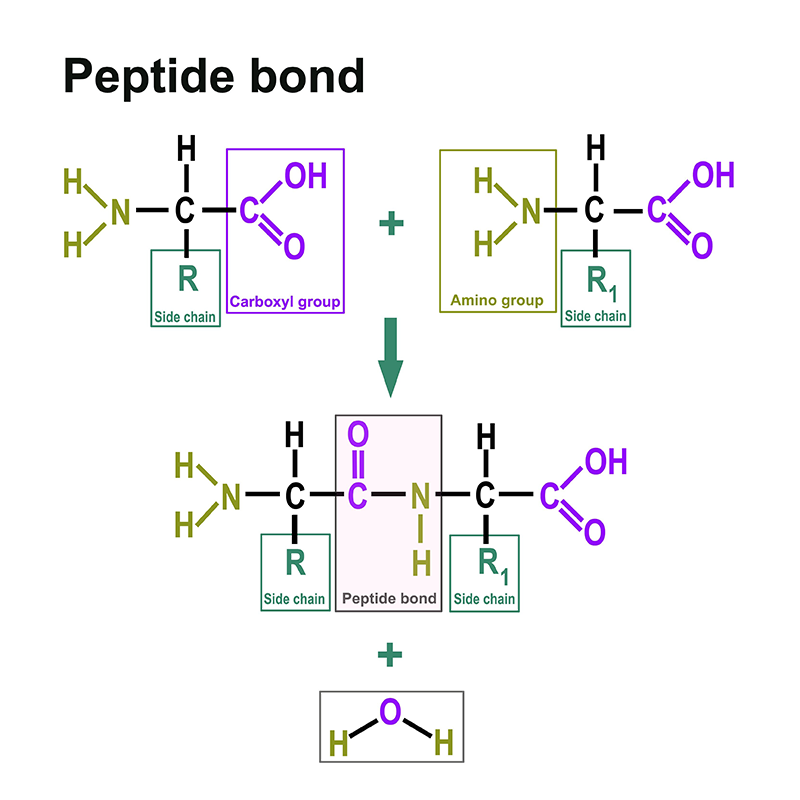
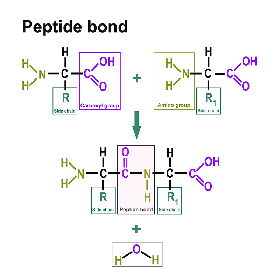
Vous trouverez une section distincte à ce sujet ici : https://www.itwreagents.com/france/fr/acides-amines. Tous les protéines sont formées d‘acides aminés reliés par des liaisons peptidiques.
Une liaison peptidique est une liaison acide carboxylique-amide qui relie deux acides aminés via le groupe carboxy d'un acide aminé et le groupe amino de l'autre acide aminé. Le plus souvent, il s'agit de la liaison entre les groupes fonctionnels respectifs en position α de deux acides aminés, c'est-à-dire entre le C-1 d'un acide aminé et le N-2 de l'autre acide aminé. Cette liaison est formée par une réaction de condensation avec élimination d'eau.
Les multiples possibilités de combinaison des acides aminés et des peptides conduisent à une possibilité presque infinie de structures et donc à de nombreuses fonctions. Les protéines peuvent être des enzymes soient des catalyseurs biologiques, des protéines de structure des cellules, de protection du matériel génétique …. Les protéines jouent un rôle déterminant dans la fonction motrice de notre corps et assurent le mouvement des muscles en actine et myosine.
L’ensemble des protéines présentes dans une cellule ou un organisme vivant dans des conditions est appelé protéome. Contrairement au génome, plutôt statique, le protéome peut changer dans sa composition protéique qualitative et quantitative en raison de conditions changeantes (facteurs environnementaux, température, expression génétique, administration de médicaments, etc.) La dynamique du protéome peut être visualisée dans l'exemple suivant. Une chenille et le papillon qui se développe à partir d'elle contiennent le même génome, mais diffèrent néanmoins extérieurement en raison d'un protéome différent. Il en va de même pour un têtard et la grenouille qui se développe à partir de lui. Les changements dans le protéome peuvent parfois se produire très rapidement, par exemple par des modifications post-traductionnelles telles que la phosphorylation et la déphosphorylation des protéines, qui jouent un rôle très important dans la transduction des signaux.
La protéomique tente de classer et de cataloguer toutes les protéines de l'organisme et de déchiffrer leurs fonctions. Les plans des protéines se trouvent dans le matériel héréditaire. Alors que le matériel génétique, l'ADN, ne fait que stocker des informations, les molécules de protéines, composées d'acides aminés, remplissent de multiples fonctions. Elles sont la substance de base de la vie et, par exemple, en tant qu'anticorps, elles préviennent les maladies, et en tant qu'enzymes, elles permettent le métabolisme et assurent le mouvement sous la forme du squelette, des tendons et des muscles.
Le mot protéome vient du chercheur australien Marc Wilkins et a été mentionné pour la première fois sur une diapositive de sa présentation au congrès "2D Electrophoresis : from protein maps to genomes" à Sienne le 5 septembre 1994. Le libellé de la diapositive était le suivant : “Proteome: the PROTEin complement expressed by a genOME, cell or tissue“ (Protéome : le complément protéique exprimé par un génome, une cellule ou un tissu).
À l'instar de la Human Genome Organisation (HUGO), les chercheurs de la International Human Proteome Organisation (HUPO) se partagent la charge de travail dans le monde entier. L'Allemagne se concentre sur la recherche sur les protéines du cerveau. En Allemagne, les principaux scientifiques spécialisés dans les protéines et la protéomique ont également uni leurs forces au sein de la Société allemande pour la recherche sur le protéome (DGPF) depuis 2001 afin d'utiliser de manière optimale les capacités de recherche.
Subdivisions
Des sous-domaines importants sont la compréhenson des interactions protéine-protéine, qui dépendent principalement des structures tertiaires et quaternaires des protéines. En outre, la purification des protéines et l'analyse quantitative de l'expression des protéines font également partie du domaine de la protéomique. Elle complète ainsi les données obtenues par l'analyse de l'expression des gènes et fournit des informations sur les composants des voies métaboliques et des circuits de régulation moléculaire. L'ingénierie des protéines permet de modifier les fonctions des protéines recombinantes pour adapter ses propriétés.
Ainsi, les techniques clés de la protéomique permettent d'élucider la fonction et la structure tridimensionnelle des protéines et d'identifier les protéines individuelles dans les mélanges.
Étant donné que tous les processus métaboliques sont effectués par des protéines, les approches thérapeutiques telles que les nouveaux agents actifs contre le cancer, les infections et certaines maladies nerveuses sont basées sur celles-ci. Des maladies telles que la drépanocytose, la maladie d'Alzheimer, la chorée de Huntington ou la maladie de Creutzfeldt-Jakob sont liées à des protéines mal formées ou/et agglutinées. Par conséquent, si l'on sait quelle protéine est responsable d'un dysfonctionnement, il devient envisageable d’avoir une action ciblée sur cette protéine pour stopper le dysfonctionnement. Les protéines recombinantes sont utilisées dans l'industrie sous la forme d'enzymes détergentes et de pesticides biologiques.
Biologie des systèmes
Un nouveau domaine de recherche qui s'appuie sur la protéomique est la biologie des systèmes. Il ne s'agit plus d'étudier uniquement les parties individuelles d'une cellule, par exemple, mais de décrire l'interaction de toutes les parties individuelles au sein d'un système et de son environnement. Outre la protéomique, cela nécessite des modèles mathématiques qui simulent le système in silico (c'est-à-dire dans des modèles informatiques), mais aussi ceux qui permettent de le reproduire dans des approches expérimentales.
Paléoprotéomique
Outre l'ADN "ancien", on peut parfois isoler des protéines fossiles à partir d'ossements fossiles, ce qui permet également de tirer des conclusions, par exemple sur leur appartenance à une espèce biologique particulière. La paléoprotéomique (du grec παλαιός palaiós, "vieux"), qui s'en inspire, bénéficie notamment du fait que certaines protéines sont plus stables que l'ADN sur une plus longue période. En 2016, par exemple, le groupe de recherche de Jean-Jacques Hublin à l'Institut Max Planck d'anthropologie évolutive a utilisé des échantillons de collagène datant de quelque 40 000 ans pour préciser que la culture archéologique de Châtelperronia est liée aux Néandertaliens et non aux humains modernes (Homo sapiens). En 2019, des protéines fossiles de la dentine de la mandibule de Xiahe découverte sur les hauts plateaux du Tibet dans la grotte de Baishiya ont prouvé qu'il s'agissait d'humains de Denisova, et quelques mois plus tard, des échantillons de dentine vieux de 1,9 million d'années ont confirmé que le genre Gigantopithecus est un taxon "frère" éteint des orangs-outans. En 2015, des analyses de collagène ont révélé une relation plus étroite entre les "ongulés d'Amérique du Sud" et les ongulés à doigts impairs, à savoir Macrauchenia et Toxodon, qui étaient encore présents à la fin du Pléistocène. Auparavant, les relations exactes entre les "ongulés d'Amérique du Sud" et les autres groupes d'ongulés n'étaient pas claires et faisaient l'objet de débats scientifiques. Pour le rhinocéros éteint Stephanorhinus, des protéomes remontant respectivement à quelque 200 000 à 400 000 et 1,8 million d'années, en 2017 et 2019, ont révélé une relation plus étroite avec le rhinocéros laineux et donc à un cercle de parenté plus étroit autour du rhinocéros de Sumatra moderne. Cette position a également pu être soutenue par des études génétiques et avait été supposée auparavant sur des bases anatomiques. Toujours en 2019, des études sur les protéines ont contribué à la reclassification systématique des paresseux fossiles et récents.
En 2015, par exemple, une étude portant sur des os vieux de 80 millions d'années de Brachylophosaurus canadensis, un membre du groupe des dinosaures à bec de canard, a attiré l'attention des médias du monde entier. L'étude a révélé des peptides qui, en raison de leur similitude avec les peptides trouvés chez les oiseaux poulets et les autruches vivant aujourd'hui, ont été interprétés comme des restes de vaisseaux sanguins.
Questions et tendances
Après quelques expériences décevantes avec des méthodes génétiques telles que l'analyse des microréseaux, certains scientifiques sont également quelque peu sceptiques à l'égard de la recherche sur le protéome. Friedrich Lottspeich, de l'Institut Max Planck de biochimie de Martinsried, président de la Société allemande pour la recherche sur le protéome (DGPF), met en garde contre les espoirs exagérés :
"Pour le secteur humain, la recherche est en fait encore trop complexe pour le moment de toute façon [...] Mais pour une analyse de la levure, qui serait un bon système modèle, là encore bien sûr personne ne veut dépenser de l'argent."
La complexité découle des nombreuses possibilités : Selon Friedrich Lottspeich, on estime que l'homme possède plusieurs centaines de milliers à des millions de protéines différentes. Un seul gène produit en moyenne cinq à dix protéines, voire plusieurs centaines dans certains cas. Appréhender pleinement cette complexité est un défi que les méthodes actuelles ne permettent pas encore de relever. D'autre part, la recherche sur le protéome se développe rapidement. Cela est dû notamment à l'amélioration constante des spectromètres de masse, qui deviennent de plus en plus précis, sensibles et rapides.
Une autre étape importante est le développement de méthodes quantitatives, telles que les procédures SILAC, iTRAQ, TMT ou ICAT basées sur l'utilisation d'isotopes stables, ou le codage métallique MeCAT, dans lequel des métaux de poids différents sont utilisés pour marquer les protéines et les peptides de différents échantillons de protéines. Ce dernier procédé permet pour la première fois dans l'approche multiplex l'utilisation à l'échelle du protéome de la spectrométrie de masse élémentaire ultrasensible (ICP-MS) (limite de détection dans la gamme des ppt à ppq inférieurs), qui permet une sensibilité plus élevée de 2 à 5 ordres de grandeur dans la quantification des protéines et présente une gamme de mesure dynamique linéaire d'au moins 6 à 8 ordres de grandeur. Contrairement aux autres méthodes qui ne quantifient "que" relativement au niveau des peptides, MeCAT permet avantageusement une quantification relative et même absolue au niveau des protéines, ce qui rend plus facile la quantification d'espèces protéiques telles que les protéines modifiées post-traductionnellement. L'étalonnage de l'ICP-MS est effectué à l'aide d'étalons métalliques indépendants des protéines/peptides. Le besoin de peptides standards spécifiques aux protéines est donc éliminé.
Si l'analyse quantitative du protéome est combinée à d'autres méthodes biologiques, il est également possible de faire des déclarations sur la fonction des protéines (par exemple, l'interaction protéine-protéine ou les modifications post-traductionnelles). La recherche moderne sur le protéome va donc désormais bien au-delà du simple catalogage des protéines et tente de comprendre des mécanismes complexes.
Applications simples
Pour la recherche, il est important de rendre les protéines visibles et de pouvoir les quantifier. L'une des techniques les plus importantes à cet égard est l'électrophorèse sur gel de polyacrylamide (PAGE). Dans la PAGE, les protéines sont séparées en fonction de leur taille dans un champ électrique. Nous vous proposons tout ce dont vous avez besoin pour préparer vous-même les gels. Vous trouverez une sélection de solutions d'acrylamide et de bisacrylamide, d'APS, de TEMED et de matériaux de soutien DTT, β-mercaptoéthanol, Coomassie populaire marqueur de protéines.
Les protéines doivent d'abord être mises à la disposition de l'observateur depuis l'extérieur. Cela se fait d'une part par des méthodes physiques, comme la perturbation des cellules par la chaleur, ou chimiques. Par exemple, notre produit Tritidy G peut aider à isoler simultanément l'ADN, l'ARN et les protéines d'un échantillon. Cependant, les détergents sont particulièrement importants dans ce contexte. De nombreux détergents bien connus comme le SDS et le Tween sont disponibles, mais aussi des détergents plus spécifiques comme la Digitonine et la Saponine, qui sont souvent utilisés pour les protéines membranaires. Nous tenons également à signaler nos produits de remplacement du Triton X-100, qui offrent une plus grande protection à l'utilisateur.
Produits de remplacement du Triton X-100 :
| CODE DU PRODUIT | NOM DU PRODUIT | NUMÉRO CAS |
| A9778 | ECOSURF™ EH-9 (remplacement du Triton X-100) |
64366-70-7 |
| A9779 | ECOSURF™ SA-9 (remplacement du Triton X-100) |
|
| A9780 | TERGITOL™ 15-S-9 (remplacement du Triton X-100) |
68131-40-8 |
L'original, le Triton X-100, est classé comme une substance à haut risque en raison du règlement REACH et n’est plus utilisable en Europe.
Les techniques de biochimie des protéines ne servent pas seulement à déterminer l'état naturel des protéines, mais aussi à produire les protéines souhaitées au moyen de manipulations ciblées. Pour ce faire, on utilise des systèmes d'expression. L'un des plus connus est l'opéron Lac. Les gènes de certaines protéines sont assemblés par biologie moléculaire à l'aide de cet opéron. Chez les bactéries (E. coli), l'organisme produit ensuite les protéines. L'ensemble du processus est induit par la substance isopropyl-β-D-thiogalactopyranoside (IPTG). Nous avons une sélection d'IPTG disponible pour tous les utilisateurs.
| CODE DU PRODUIT | NOM DU PRODUIT | NUMÉRO CAS |
| A1008 | IPTG BioChemica | 367-93-1 |
| A4773 | IPTG pour la biologie moléculaire, sans dioxane | 367-93-1 |
| A7211 | IPTG d'origine végétale galactose, sans dioxane | 367-93-1 |
Le plus grand risque de dégradation d'une protéine est ... une autre protéine. La dégradation est catalysée par des protéases. Celles-ci sont présentes dans tous les organismes et donc souvent aussi dans les échantillons des utilisateurs/scientifiques. Comme il serait souvent peu rentable de réaliser une séparation directe, on utilise des inhibiteurs.
Ceux-ci interfèrent avec l'activité catalytique des protéases de sorte qu'elles ne peuvent pas devenir actives et que les autres enzymes restent intactes.
Parfois, cependant, c'est précisément la dégradation des protéines qui est souhaitée. Par exemple, si l'on veut examiner uniquement les acides nucléiques d'un échantillon. Les protéines peuvent alors constituer une source d'interférence. Un exemple bien connu est celui de certains kits de test pour le COVID-19. La protéinase K est une protéase qui catalyse la dégradation des protéines. Nous proposons la protéinase K de haute qualité sous forme lyophilisée et déjà dissoute, prête à l'emploi.
D'autres produits importants qui sont utilisés sont les albumines et les cofacteurs tels que le NAD et le NADPH. Ils peuvent être nécessaires comme stabilisateurs ou comme coenzymes dans diverses réactions.
Le domaine de la protéomique est passionnant. Il nous montre le véritable état réel de chaque cellule. De nouveaux axes de recherche conduiront encore à des découvertes révolutionnaires dans ce domaine.
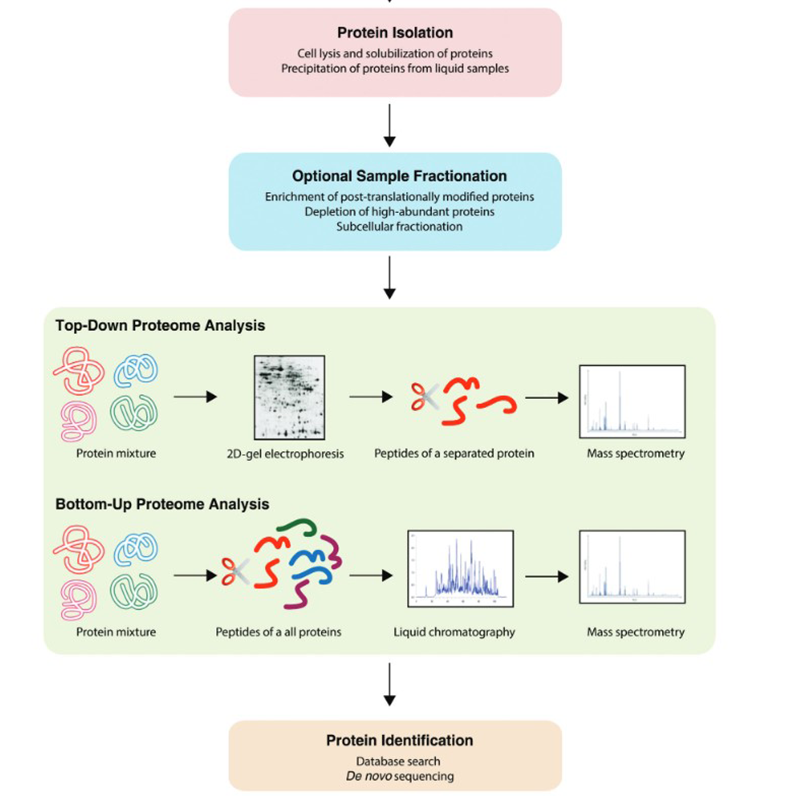
Outre les techniques déjà mentionnées, on trouve différents flux de travail dans le domaine de la protéomique. Elles peuvent avoir lieu de bas en haut ou de haut en bas :
.png)
La protéomique est également réalisée au moyen de modèles dans lesquels on utilise des modèles cellulaires ou animaux. L'électrophorèse sur gel 2D et le marquage jouent un rôle. Les grandes étapes dans le domaine de la protéomique ont été réalisées par des méthodes chromatographiques couplées à la spectrométrie de masse. Nous proposons les solvants les plus courants pour ces applications.
Les méthodes couramment utilisées aujourd'hui en protéomique comprennent la chromatographie, la spectrométrie de masse, l'imagerie MALDI, l'électrophorèse unidimensionnelle sur polyacrylamide (1D PAGE), l'électrophorèse bidimensionnelle sur gel de polyacrylamide (2D PAGE), les microréseaux, l'analyse des acides aminés (AAA), la culture cellulaire et, surtout, une large utilisation des modèles bioinformatiques.
La protéomique qualitative et quantitative est notamment utilisée pour le diagnostic des tumeurs et permet de tirer des conclusions sur le pronostic et les thérapies de chaque patient.
Les protéines ont également une grande importance en immunologie. Nos anticorps sont des formes particulières de protéines.
La complexité de la protéomique découle des très nombreuses possibilités : Selon Friedrich Lottspeich (biochimiste autrichien), on estime que l'homme possède plusieurs centaines de milliers à des millions de protéines différentes. Un seul gène produit en moyenne cinq à dix protéines, dans certains cas plusieurs centaines. Saisir pleinement cette complexité est un défi que les méthodes actuelles ne permettent pas encore de relever.
Vous trouverez ci-dessous un aperçu des articles importants, adaptés à la recherche en biochimie des protéines (protéomique) :
| CODE DU PRODUIT | NOM DU PRODUIT | NUMÉRO CAS |
| A1088 | ABTS® BioChemica | 30931-67-0 |
| A4983 | Acrylamide solution (30%) - Mélange 29 : 1 pour biologie moléculaire | |
| A3626 | Acrylamide solution (30%) - Mélange 37.5 : 1 pour biologie moléculaire | |
| A3658 | Acrylamide solution (40%) - Mélange 19 : 1 pour biologie moléculaire | |
| A0385 | Acrylamide solution (40%) - Mélange 29 : 1 pour biologie moléculaire | |
| A4989 | Acrylamide solution (40%) - Mélange 37.5 : 1 pour biologie moléculaire | |
| A7582 | Acrylamide solution 2K (18%) pour la dénaturation de l'ADN-PAGE | |
| A7590 | Acrylamide solution 2K (8%) pour la dénaturation de l'ADN-PAGE | |
| A1089 | Acrylamide 2K standard grade, extrapure | 79-06-1 |
| A0951 | Acrylamide solution 4K (30%) - Mélange 29 : 1 | |
| A1672 | Acrylamide solution 4K (30%) - Mélange 37,5:1 | |
| A1577 | Acrylamide solution 4K (40%) - Mélange 37,5:1 | |
| A1090 | Acrylamide 4K ultrapure | 79-06-1 |
| A1421 | AEBSF Chlorhydrate BioChemica | 30827-99-7 |
| A0850 | Albumine (BSA) grade EIA et RIA | 9048-46-8 |
| A2244 | Albumine (BSA) Fraction V (pH 5,2) | 9048-46-8 |
| A1391 | Albumine (BSA) Fraction V (pH 7,0) | 9048-46-8 |
| A6588 | Albumine (BSA) Fraction V (pH 7,0) pour le transfert Western | 9048-46-8 |
| A4344 | Albumine brute de blanc d'œuf de poule | 9006-59-1 |
| A1523 | 4-Aminoantipyrine BioChemica | 83-07-8 |
| A7708 | AppliCoat Plate Stabilizer | |
| A2132 | Aprotinine BioChemica | 9087-70-1 |
| A2568 | Avidin BioChemica | 1405-69-2 |
| A1117 | BCIP BioChemica | 6578-06-9 |
| A3636 | Bisacrylamide pour biologie moléculaire | 110-26-9 |
| A3417 | CheLuminate-HRP PicoDetect | |
| A2144 | Chymostatine | 9076-44-2 |
| A1101 | DTT BioChemica | 3483-12-3 |
| A2948 | DTT pour biologie moléculaire | 3483-12-3 |
| A3668 | DTT solution 1 mol/L (1 M) pour biologie moléculaire | 3483-12-3 |
| A1007 | X-Gal BioChemica | 7240-90-6 |
| A4978 | X-Gal pour biologie moléculaire | 7240-90-6 |
| A2243 | L-Glutathion oxydé BioChemica | 27025-41-8 |
| A9782 | L-Glutathion réduit (USP) pur, qualité pharma | 70-18-8 |
| A2084 | L-Glutathion réduit BioChemica | 70-18-8 |
| A1828 | Inhibiteur de trypsine de soja > 7000 BAEE | 9035-81-8 |
| A1008 | IPTG BioChemica | 367-93-1 |
| A4773 | IPTG pour biologie moléculaire, sans dioxane | 367-93-1 |
| A7211 | IPTG de galactose d'origine végétale, sans dioxane | 367-93-1 |
| A0830 | Lait en poudre écrémé | |
| A2185 | Luminol | 521-31-3 |
| A8889 | Marqueur protéique VI (10 - 245) pré-coloré | |
| A1108 | β-Mercaptoéthanol pour biologie moléculaire | 60-24-2 |
| A1243 | NBT BioChemica | 298-83-9 |
| A1272 | 2-Nitrophényl-β-D-Galactopyranoside BioChemica | 369-07-3 |
| A1028 | 4-Nitrophényl-β-D-Glucuronide BioChemica | 10344-94-2 |
| A2205 | Pepstatine A | 26305-03-3 |
| A0999 | PMSF BioChemica | 329-98-6 |
| A2935 | Ponceau S - Solution | |
| A1495 | Streptavidine ultrapure | 9013-20-1 |
| A1148 | TEMED | 110-18-9 |
| A3840 | 3,3',5,5'-Tétraméthylbenzidine BioChemica | 54827-17-7 |