ITW Reagents se enorgullece en presentar la tecnología DNA-ExitusPlusTM
DNA-ExitusPlusTM & DNA-ExitusPlusTM IF
Reactivo para la eliminación de contaminaciones de ADN y ARNCódigos de producto A7089 y A7409
Publicaciones disponibles:
InfoPoint
Product-Info
Folleto
Póster para laboratorio (solicita tu póster a tu distribuidor local)
Descripción
DNA-ExitusPlus™ es un reactivo patentado para la eliminación de contaminaciones de ADN y ARN de superficies y equipos de laboratorio (1). La solución emplea una química suave y no corrosiva para una rápida degradación no enzimática de los ácidos nucleicos. Los ya cortos tiempos de incubación con el ADN-ExitusPlus™ eliminan completamente el ADN y el ARN no deseados de las superficies de trabajo y las herramientas (1, 2).Hay dos versiones diferentes de DNA-ExitusPlus™ disponibles: DNA-ExitusPlus™ (A7089) incluye un indicador de color para visualizar fácilmente la superficie cubierta con el reactivo. DNA-ExitusPlus IF™ (A7409) es casi sin color. Ambas soluciones se oscurecen con el tiempo debido a los componentes redox-activos contenidos en las soluciones.
Nota: No hay diferencias en los protocolos de aplicación de DNA-ExitusPlus™ y DNA-ExitusPlus IF™! Por lo tanto, no nombramos la forma IF en las siguientes instrucciones de uso.
Las características únicas de DNA-ExitusPlus™

- Los efectos catalíticos y cooperativos de los componentes causan una degradación muy rápida no enzimática y no específica de la secuencia de las moléculas de ADN y ARN que hace desaparecer toda contaminación.
- Todos los componentes del DNA-ExitusPlus™ son fácilmente biodegradables y no son dañinos o tóxicos para los humanos.
- No se utilizan ácidos minerales o sustancias alcalinas agresivas. El equipo y los materiales no se dañan o corroen incluso después de un tiempo de incubación prolongado.
- No hay gases tóxicos.
- Las temperaturas elevadas por encima de los 50°C aceleran la reacción y la actividad.
Instrucciones de uso
- El tiempo de incubación óptimo para la descontaminación de las superficies es de 10 minutos y a una temperatura superior a 20°C. Después de la incubación, el DNA-ExitusPlus™ residual se limpia con un papel absorbente. No es necesario limpiar adicionalmente con agua estéril posteriormente. Esta es una nueva ventaja en comparación con las soluciones de descontaminación tradicionales.
- Después de que la solución se haya secado completamente, no hay más reacción de descontaminación. Por lo tanto, un tiempo de incubación de más de 30 minutos no es necesario ni útil. En caso de contaminaciones severas se recomienda una segunda aplicación de la solución para una mayor eficiencia.
- Para la eliminación de los rastros residuales no deseados y secos del reactivo, recomendamos eliminar estos rastros con agua estéril o con un tampón 10X TE y un papel absorbente.
Para descontaminar las superficies del laboratorio: Aplicar DNA-ExitusPlus™ directamente sobre la superficie del laboratorio. Incubar durante 10 minutos. Limpie bien los residuos de DNA-ExitusPlus™ con una toalla de papel (elimine los residuos secos con agua estéril / tampón TE 10X). No es necesario aclarar con agua.
Para descontaminar los aparatos de laboratorio: Aplique generosamente DNA-ExitusPlus™ en una toalla de papel y limpie bien todas las superficies expuestas del aparato. Secar con una toalla de papel limpia. Para limpiar piezas pequeñas, sumérjalas brevemente en DNA-ExitusPlus™ y séquelas.
Para descontaminar los recipientes de plástico y vidrio: Añada una cantidad suficiente de DNA-ExitusPlus™ para permitir el recubrimiento de toda la superficie del recipiente mediante agitación o vórtice. Deseche la solución y séquela. Enjuague bien los recipientes con agua destilada y séquelos.
Para descontaminar las pipetas: Siguiendo las instrucciones del fabricante, retire el eje de la pipeta y retire los sellos y las juntas del eje. Sumerja el eje durante un minuto en DNA-ExitusPlus™, enjuague bien el eje con agua, deje secar y vuelva a montarlo.
Una nueva tecnología para la descontaminación de ácidos nucleicos
La amplificación del ADN es una de las técnicas más utilizadas en el laboratorio de investigación moderno. La presencia de ADN contaminante en las estaciones de trabajo de la PCR y sus alrededores puede provocar artefactos no deseados durante la amplificación. Principalmente, hay dos maneras de hacer que el ADN no sea amplificable:- por degradación del ADN (por ejemplo, mediante la adición de DNasas o la destrucción química), o
- por modificación de las bases, dejando la cadena de ADN intacta, pero bloqueada para su lectura por las polimerasas.
Utilizando un ensayo de rotura de hebras de ADN (diseñado por multiBIND biotec GmbH, Alemania; véase la Fig. 1), se ha demostrado que no todas las soluciones de descontaminación de ADN del mercado degradan totalmente el ADN. DNA-ExitusPlus™ supone una mejora respecto a esos productos y provoca tanto la rotura de la cadena como la degradación. Cuando se utiliza correctamente en el área de trabajo, elimina totalmente la amplificación del ADN no objetivo (Fig. 3). DNA-ExitusPlus™ es una solución de limpieza no alcalina, no corrosiva y no cancerígena que es eficaz en todas las superficies.
Una grave desventaja de los reactivos de descontaminación convencionales se pone de manifiesto en una nueva prueba sobre el potencial corrosivo de sus componentes. Para ello, se incubaron diferentes placas de metal durante 20 minutos con alícuotas idénticas de los reactivos. Los metales seleccionados son representativos de los equipos y materiales que se encuentran en los laboratorios. El resultado de esta prueba de corrosión se documenta en la figura 2, mostrando que todos los productos comerciales conocidos contienen sustancias químicas agresivas con efectos corrosivos, nocivos o incluso tóxicos. Se sabe que estos reactivos convencionales contienen azidas, ácidos minerales como el ácido fosfórico o el ácido clorhídrico, peróxidos agresivos o sustancias alcalinas fuertes como el hidróxido de sodio. Incluso tras una incubación de sólo 20 minutos, se observan en muchos casos daños irreversibles en las superficies metálicas (véase la figura 2). La solución recientemente desarrollada DNA-ExitusPlus™ muestra sus características únicas especialmente en esta prueba de corrosión. En todas las superficies metálicas ensayadas, no se observa ningún daño o corrosión después del tratamiento. DNA-ExitusPlus™ también fue probado en condiciones idénticas en muchas superficies de plástico diferentes sin ninguna indicación de daño (datos no mostrados).
El reactivo es estable durante al menos 12 meses y es estable al calor.
(1) Esser, K.-H. et al. (2006) DNA decontamination: DNA-ExitusPlus™ in comparison with conventional reagents. Nature Methods 3, 151
(2) Arena, A. (2010) DNA-ExitusPlus™ versus standard bleach solution for the removal of DNA contaminants on work surfaces and tools. Investigative sciences journal 2, 20-29
Control de calidad
Cuantificación de la degradación del ADN mediante electroforesis analítica en gel de agarosa o PCRCada vez es más importante controlar con exactitud la degradación del ADN tras las medidas de descontaminación en los laboratorios.
Si se incuba el ADN con DNA-ExitusPlus™ o cualquier otro reactivo de descontaminación y se carga una muestra de la mezcla de reacción en un gel de agarosa sin neutralización o desnaturalización previas, no será posible una determinación cuantitativa de la degradación del ADN. Muchos fragmentos se pegarán para formar unidades más grandes, incluso después de la rotura de la hebra (10 a 20 pares de bases homólogas serán suficientes para hibridar formando unidades más grandes). Este fenómeno de la hibridación se observa para los fragmentos de ADN con extremos pegajosos, por ejemplo, del marcador de tamaño de ADN λ para la electroforesis en gel. Esta es la razón por la que dicho ADN marcador se desnaturaliza antes de cargarlo en un gel.
Los reactivos de otros proveedores suelen contener altas concentraciones de ácidos o bases fuertes. Si no se neutralizan estas mezclas de reacción antes de cargarlas en el gel, se observará un cambio en el color del indicador de pH azul de bromofenol (intervalo de transición a pH 2 - 4,6 de verde-amarillo a azul-violeta). Si carga una muestra no neutralizada en un gel, podrá observar la destrucción de la ranura por los productos químicos. El bromuro de etidio se destruirá en estas condiciones y, por tanto, no puede aplicarse, ya que no tiñe el ADN. Los carriles del gel serán claros, aunque haya grandes cantidades de ADN sin teñir. Neutralizando la muestra con tampón Tris, se hará visible el color correcto del azul de bromofenol. Dependiendo de la composición del reactivo de descontaminación, la neutralización debe realizarse con 100 mM Tris pH 12 o 100 mM Tris pH 3, respectivamente. En el caso del DNA-ExitusPlus™, la capacidad tampón del tampón de carga es suficiente para tamponar incluso una mezcla 1:1 del reactivo de descontaminación y la muestra.
Cuando la neutralización del azul de bromofenol muestra el pH correcto, las muestras se desnaturalizan a 90°C durante 2 minutos.
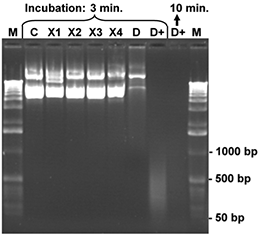
Alícuotas de un ADN plasmídico CCC (7 kb), 200 ng para cada muestra, se disolvieron en 10 μl de agua y se trataron con 5 μl de uno de los reactivos enumerados durante 3 o minutos, respectivamente, a temperatura ambiente. Por último, las muestras se mezclaron con tampón de carga de azul de bromofenol y se desnaturalizaron durante 3 minutos a 92 °C. Las muestras desnaturalizadas se colocaron en hielo y la mezcla de reacción completa se cargó en un gel de agarosa al 1 %. Tras la electroforesis en gel, el gel de agarosa se tiñó con bromuro de etidio y se fotografió. El control (C) contiene el ADN del plásmido CCC intacto (200 ng) tras el tratamiento con 5 µl de agua estéril. Los cortes y daños en las hebras de ADN generan fragmentos de menor peso molecular. Estos fragmentos de ADN más pequeños pueden identificarse en el gel por comparación con la muestra de control y el marcador de peso molecular (M; 1 kb ladder). Los productos X1-X4 causan muy poca degradación del ADN de prueba solamente, mientras que el producto D (DNA-Exitus™ convencional) fue más eficaz en estas condiciones. Sólo el DNA-ExitusPlus™ (D+) causa una degradación muy rápida y casi completa del ADN después de 3 minutos. De este modo, sólo se observan fragmentos residuales de ADN menores de 500 pares de bases. La incubación prolongada (10 min.) destruyó todo el ADN plasmídico.
La alta concentración de productos químicos en los reactivos de otros proveedores provoca graves problemas durante el análisis de la PCR. Las muestras recogidas por la limpieza de los bancos tratados con dichos reactivos contenían sustancias químicas en concentraciones tan altas que la PCR se inhibía, incluso después de la dilución. Por lo tanto, se requiere un control positivo de la PCR con una mezcla de una plantilla definida y una alícuota de la muestra recogida para excluir resultados falsos negativos. En nuestras manos, dicho control negativo-PCR seguía conteniendo ingredientes de los reactivos en concentraciones inhibidoras que hacían necesaria una nueva dilución o neutralización.
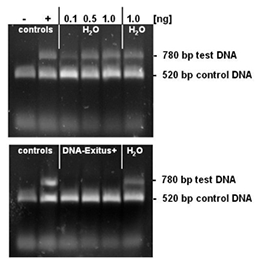
Se liofilizaron cantidades seleccionadas (de 0,1 a 1 ng) de un ADN de prueba en la superficie interior de los tubos de PCR. Los tubos de PCR con la muestra de ADN liofilizada se incubaron durante 20 segundos con agua estéril o con DNA-ExitusPlus™ . A continuación, los tubos se lavaron dos veces con 100 µl de agua estéril. Para la reacción de prueba PCR, se añadieron mezclas de 50 µl a cada tubo. Esta mezcla de reacción contiene cebadores para la amplificación del ADN de control (control) y del ADN de prueba. El ADN de control (1 ng) se añade en cada muestra y se comprueba que la reacción de PCR no se inhibe. La amplificación de una banda de ADN correspondiente al ADN de prueba indica que todavía hay moléculas de ADN intactas de esta plantilla. Tras la degradación completa y la eliminación del ADN de prueba, la reacción de PCR no debería amplificar ningún fragmento de ADN para esta plantilla. Tras la electroforesis en un gel de agarosa al 1%, el gel se tiñó con bromuro de etidio y se documentó. El control negativo con agua estéril (H2O) exhibe bandas de ADN para el patrón de ensayo y de control. La reacción de PCR tras el tratamiento con DNA-ExitusPlus™ amplifica únicamente el fragmento del ADN de control. Esto demuestra que el tratamiento con DNA-ExitusPlus™ destruye y elimina todo rastro de ADN del patrón de ensayo.

Para esta prueba se eligieron placas metálicas típicas de los materiales y equipos de laboratorio. Se aplicaron alícuotas de 10 μl de cada reactivo de la lista sobre las diferentes superficies metálicas. Se utilizó agua estéril como control (0). Tras un tiempo de incubación de 20 minutos, se limpiaron los reactivos y se lavaron brevemente los metales con agua estéril. Tras el secado completo, se fotografiaron las placas metálicas. Los reactivos X2, X3 y D (véase la figura 2) para la descontaminación del ADN causan corrosión y daños irreversibles en muchas de las superficies metálicas. En el caso de DNA-ExitusPlus™ (D+) no se observan daños en ninguna de las superficies. En algunos casos se observa un efecto de pulido por la eliminación de suciedad o capas de óxido.
Degradación de pequeños fragmentos de ADN
La actividad de ruptura de la cadena del DNA-ExitusPlus™ es independiente del tamaño de los fragmentos de ADN, ya que la destrucción se basa en una acción química y no en una actividad enzimática. Por lo tanto, se ha incubado un producto de PCR de 750 pb durante distintos tiempos con DNA-ExitusPlus™ (véase la Fig. 4). Como era de esperar, los cebadores no pueden detectarse ni siquiera después del tiempo de incubación más corto. Tras 5 minutos de incubación con DNA-ExitusPlus™ se destruye casi todo el ADN. Queremos aclarar por qué es así: Suponiendo una actividad de mellado teórica de 100.000 mellas por minuto, todos los fragmentos de ADN serán destruidos, independientemente de su tamaño. Los fragmentos más pequeños desaparecerán primero que los más grandes. Por lo tanto, trasladando esta teoría a una molécula de prueba (forma ccc, plásmido de 6 kb), después de 5 minutos quedará una pequeña fracción de fragmentos con un tamaño de 200 a 500 pb solamente. Las mellas se introducirán estadísticamente en cualquier sitio, sin dejar una sola clase de fragmentos. Por lo tanto, el análisis PCR será negativo. Al pulverizar DNA-ExitusPlus™ sobre superficies en el laboratorio, se aplicará un enorme exceso de aproximadamente 1 a 5 ml de reactivo a las cantidades más pequeñas de ADN.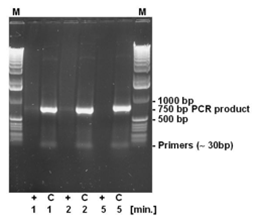
Para comprobar la degradación de pequeños fragmentos de ADN, se han incubado 500 ng de ADN de una reacción de PCR que produce un fragmento de PCR de 750 pb con DNA-ExitusPlus™ durante los períodos indicados (1, 2 y 5 minutos). Tras la incubación, el ADN se desnaturalizó a 95 °C durante 2 minutos. (+) 5 µl de ADN más 5 µl de DNA-ExitusPlus™ ; (C) control 5 µl de ADN más 5 µl de agua; (M) marcador de peso molecular 1 kb ladder.
Degradación del ARN con DNA-ExitusPlus™
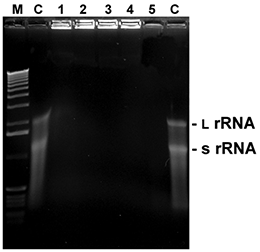
Fig. 5. Degradación del ARN
Se mezclaron 5 µl con 1 µl de ARN total de E. coli con 5 µl de los reactivos indicados y se incubaron a RT durante el tiempo indicado. A continuación, las muestras se mezclaron con tampón de carga, se calentaron durante 2 minutos a 60°C y se cargaron inmediatamente en un gel de agarosa al 1,5% con formamida/formaldehído como agentes desnaturalizantes. Tras el lavado y la renaturalización del gel, el ARN se visualizó mediante tinción con EtBr (ARNr L: ARN ribosómico grande; ARNr s: ARN ribosómico pequeño).
M: 1 kb ladder
C: control (agua estéril)
1:D+ 0,5 minutos
2:D+ 1 minuto
3:D+ 2 minutos
4:D+ 5 minutos
5: RNasaA (10 ng / 5 minutos)
Aumento de la eficacia mediante el incremento de la temperatura de incubación
Es extremadamente difícil eliminar y destruir el ADN, desecado en cualquier superficie o el ADN "protegido" por, por ejemplo, la envoltura de un virus. Incluso la esterilización en autoclave no consigue en algunos casos degradar dicho ADN. El genoma completo de un virus aviar pudo ser detectado después del autoclave [Elhafi, G., Naylor, C.J., Savage, C.E. y Jones, R.C. (2004). Los tratamientos en microondas o en autoclave destruyen la infectividad del virus de la bronquitis infecciosa y del neumovirus aviar, pero permiten su detección mediante la reacción en cadena de la polimerasa con transcriptasa inversa. Avian Pathology 33, 303-306]. Se ha probado la actividad de DNA-ExitusPlus™ como aditivo de las soluciones durante el autoclave. ¡Se pudo demostrar, que el aumento de las temperaturas mejoró la actividad de degradación del ADN de DNA-ExitusPlus™ !
Cultivos de 50 ml de cepas recombinantes de E. coli fueron esterilizados en autoclave a 120°C y 1,2 bares durante 20 minutos con la adición de volúmenes iguales de agua (-) o de DNA-ExitusPlus™ (+). Posteriormente, se investigaron alícuotas de 10 µl de estos cultivos mediante geles de agarosa de ADN analítico. La muestra con agua estéril añadida (-) muestra grandes cantidades de fragmentos de ADN de alto peso molecular. La adición del mismo volumen de DNA-ExitusPlus™ (+) provoca la degradación del ADN, mostrando tamaños de fragmentos inferiores a 20 pares de bases.

Los cultivos recombinantes de E. coli esterilizados en autoclave contienen un plásmido con el gen de resistencia a la ampicilina (AmpR-Gen). Se analizaron alícuotas (2 µl) de los cultivos en una reacción de PCR con cebadores para el gen AmpR completo después de la esterilización en autoclave. La muestra con agua estéril (-) da lugar a una banda fuerte del gen AmpR completo (930 pb), mientras que las muestras tratadas con DNA-ExitusPlus™ (+) evitaron un resultado positivo. Como control positivo (K), se mezcló una alícuota de 2 µl de la muestra con DNA-ExitusPlus™ con 2 ng de ADN molde que codifica el gen AmpR. La amplificación de la banda correspondiente muestra que la reacción de PCR sí funciona en las condiciones del ensayo.
FAQs
¿Cuál es el tiempo de contacto sugerido?Para contaminaciones con pequeñas cantidades de ADN son suficientes de 5 a 10 minutos a temperatura ambiente. Para cantidades mayores de ADN se recomienda repetir el procedimiento después de limpiar el DNA-ExitusPlus™ .
Además, el aumento de la temperatura de la solución a 50°C o 60°C aumenta sustancialmente la tasa de degradación del ADN.
¿Existe algún método para detectar los residuos de los materiales componentes de DNA-ExitusPlus™?
La nueva formulación de DNA-ExitusPlus™ contiene una baja cantidad de indicador de color que puede detectarse en las superficies después del secado. En caso de que haya que eliminar cualquier pequeño rastro residual de DNA-ExitusPlus™, se puede limpiar fácilmente de las superficies con papel de filtro empapado en agua estéril.
Para la inactivación de mayores cantidades residuales de DNA-ExitusPlus™, las superficies pueden tratarse con una solución de tampón TE 10X estéril, pH 8,0. Asegúrese de evitar nuevas contaminaciones durante este tratamiento.
Su equipo incluye juntas de polipropileno, acero inoxidable, EPDM y silicona. ¿Qué información tenemos en cuanto al "recubrimiento" o adherencia de estos materiales?
EPDM = Etileno-Propileno-Dieno-Monómero
El polipropileno, el acero inoxidable, el EPDM y las juntas de silicona no resultan dañados por DNA-ExitusPlus™ y la solución puede lavarse fácilmente con agua estéril o con tampón TE 10X, pH 8,0. Las cantidades residuales de DNA-ExitusPlus™ se han secado en estas superficies y pueden eliminarse fácilmente con un solo paso de lavado con las mismas soluciones indicadas y limpiando la solución con toallas de papel limpias.
Tubos finos: ¿cómo puedo eliminar los residuos de DNA-ExitusPlus™?
DNA-ExitusPlus™ tiene la ventaja sobre otros productos comerciales especialmente para los tubos con un diámetro pequeño, porque la fuerza iónica de todos los componentes son sustancialmente más bajos que para otros productos comerciales.
Además, todos los componentes de DNA-ExitusPlus™ son altamente solubles en agua y no cambian la viscosidad del agua sustancialmente. Todos ellos tienen una afinidad muy baja por las superficies de metal o plástico.
Por lo tanto, incluso para los tubos con un diámetro muy pequeño, un paso de enjuague con tampón TE 10X, pH 8,0, y un enjuague final con agua estéril es suficiente para la eliminación completa de DNA-ExitusPlus™ .
Como control final, determine el pH del agua estéril después del enjuague final. Un pH entre 6 y 8 es aceptable. Por favor, compruebe el pH del agua estéril antes de la aplicación. Precaución: el agua estéril preparada con resinas de intercambio aniónico puede presentar un pH bajo.
¿Por qué se observa una fluorescencia brillante en las ranuras de las muestras tratadas con DNA-ExitusPlus™ de la fig. 5?
La fluorescencia que se observa en las ranuras del gel de agarosa está causada por los ingredientes del ADN-ExitusPlus™ residual, pero no por el ADN o el ARN. Estas pequeñas moléculas se difunden en todas las direcciones en la matriz del gel a pesar de la electroforesis. Por el contrario, el ADN se acumularía en el fondo de la ranura, en caso de precipitarse. En las condiciones experimentales elegidas, ni el ARN ni el ADN precipitarían y las muestras de ARN de este experimento no contienen ADN de alto peso molecular retenido en las ranuras.