Prodotti sponsorizzati
-

PMSF BioChemica
A0999 -

IPTG BioChemica
A1008 -

DTT BioChemica
A1101 -

NBT BioChemica
A1243 -

-

107 prodotti disponibili
- 2-Nitrofenil-ß-D-Galattopiranoside BioChemica A1272,0025 · 369-07-3
- 3,3',5,5'-Tetrametilbenzidina BioChemicaA3840,0025 · 54827-17-7
- 4-Aminoantipirina BioChemica A1523,9015 · 83-07-8
- ABTS® BioChemica A1088,0005 · 30931-67-0
- ABTS® BioChemica A1088,0010 · 30931-67-0
- Acrilammide - Soluzione (30 %) - Miscela 29 : 1 per biologia molecolareA4983,0500 ·
- Acrilammide - Soluzione (30 %) - Miscela 37.5 : 1 per biologia molecolareA3626,1000 ·
- Acrilammide - Soluzione (30 %) - Miscela 37.5 : 1 per biologia molecolareA3626,0500 ·
- Acrilammide - Soluzione (40%) - Miscela 19 : 1 per biologia molecolareA3658,1000 ·
- Acrilammide - Soluzione (40%) - Miscela 29 : 1 per biologia molecolareA0385,1000 ·
- Acrilammide - Soluzione (40%) - Miscela 29 : 1 per biologia molecolareA0385,0500 ·
- Acrilammide - Soluzione (40%) - Mix 37.5 : 1 per biologia molecolareA4989,0500 ·
- Acrilammide 2K Grado standard, puro A1089,0500 · 79-06-1
- Acrilammide 4K - Soluzione (30 %) - Mescolare 37.5 : 1 A1672,1000 ·
- Acrilammide 4K - Soluzione (30 %) - Mix 29 : 1 A0951,1000 ·
Biochimica delle proteine
Le proteine si trovano in ogni cellula e la loro sequenza è già mappata dal codice genetico. L’informazione contenuta nei geni viene tradotta in proteine nel corso di un processo chiamato espressione genica, in modo che le proteine giuste siano disponibili al momento giusto nell'essere vivente.
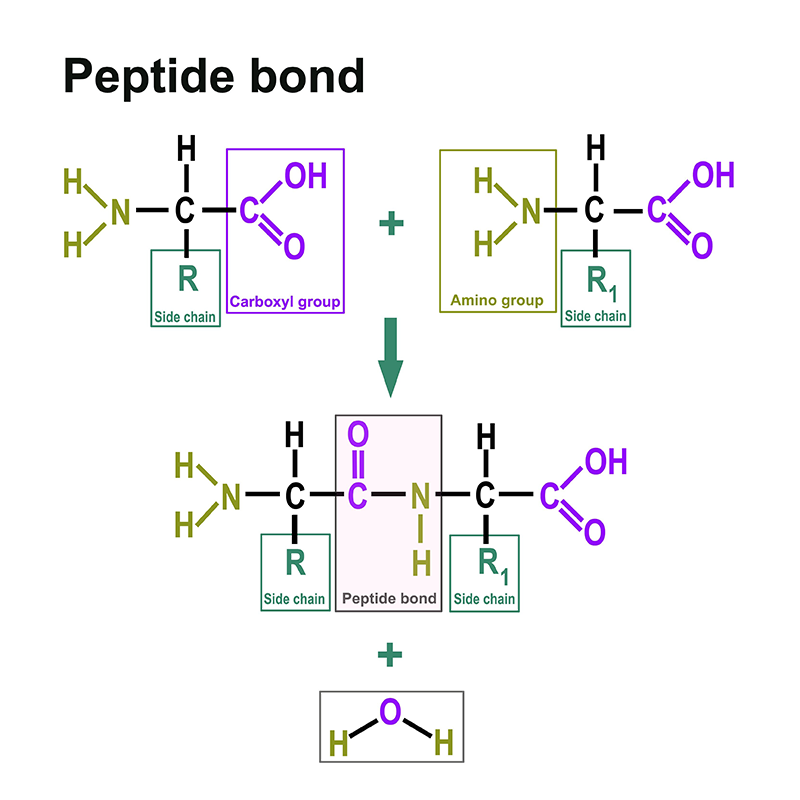
Le proteine sono costituite da aminoacidi. Puoi trovare una sezione separata al seguente link: https://www.itwreagents.com/italy/it/aminoacidi. Gli aminoacidi sono legati fra loro attraverso il legame peptidico.
Il legame peptidico è un legame covalente che collega due aminoacidi attraverso il gruppo carbossilico di un aminoacido e il gruppo amminico dell'altro aminoacido. Più comunemente, si intende il legame tra i rispettivi gruppi funzionali in posizione α di due aminoacidi, cioè tra il C-1 di un aminoacido e l'N-2 dell'altro aminoacido. Il legame peptidico si genera tramite una reazione di condensazione con eliminazione di una molecola d’acqua.
Le molteplici possibilità di combinazione degli aminoacidi e anche dei peptidi portano a una possibilità quasi infinita di strutture e quindi a molte funzioni differenti. Le proteine possono essere enzimi e quindi i catalizzatori biologici. Le proteine possono dare struttura alle nostre cellule, proteggere il nostro materiale genetico e mantenerlo protetto. Le proteine giocano un ruolo decisivo nella funzione motoria del nostro corpo e sono causa del movimento dei nostri muscoli (actina e miosina). L'insieme delle proteine di un organismo prende il nome di proteoma.
Più precisamente, il proteoma comprende l'insieme di tutte le proteine presenti in una cellula o in un organismo vivente in condizioni definite e in un tempo definito. In contrasto con il genoma piuttosto statico, il proteoma e anche il trascrittoma sono dinamici e possono quindi cambiare nella loro composizione proteica qualitativa e quantitativa a causa del cambiamento delle condizioni (fattori ambientali, temperatura, espressione genica, somministrazione di farmaci, ecc.). La dinamicità del proteoma può essere visualizzata nel seguente esempio: Un bruco e la farfalla, che sda esso si sviluppa, hanno lo stesso genoma ma differiscono esternamente a causa di un proteoma diverso. Lo stesso vale per un girino e la rana che si sviluppa da esso.
I cambiamenti nel proteoma possono avvenire a volte molto rapidamente, per esempio attraverso modifiche post-traslazionali come la fosforilazione e la defosforilazione delle proteine, le giocano un ruolo molto importante nella trasduzione dei segnali all’interno della cellula.
La proteomica cerca di classificare e catalogare tutte le proteine dell'organismo e di decifrare le loro funzioni. Le cianografie delle proteine si trovano nel materiale ereditario. A differenza del DNA, che immagazzina solo informazioni, le molecole proteiche svolgono molteplici compiti. Sono la sostanza di base della vita e hanno molteplici funzioni ad esempio, come anticorpi prevengono le malattie, come enzimi permettono il metabolismo.
La parola proteoma viene dal ricercatore australiano Marc Wilkins e fu menzionata per la prima volta su una diapositiva nella sua presentazione al congresso "Elettroforesi 2D: dalle mappe proteiche ai genomi" a Siena il 5 settembre 1994. La dicitura sulla diapositiva era: “Proteome: the PROTEin complement expressed by a genOME, cell or tissue” (Proteoma: il complemento di PROTEine espresso da un genOMA, cellula o tessuto).
Come l'Organizzazione del Genoma Umano (The Human Genome Organisation, HUGO), i ricercatori dell'Organizzazione Internazionale del Proteoma Umano (HUPO) condividono il loro lavoro in tutto il mondo. La Germania ad esempio si sta concentrando sulla ricerca sulle proteine del cervello. In Germania, gli scienziati nel di maggio rilievo nel settore delle proteine e della proteomica hanno anche unito le loro forze nella Società tedesca per la Ricerca sul Proteoma (DGPF) dal 2001, al fine di ottimizzare e avanzare insieme nelle scoperte scientifiche.
Suddivisioni
Alcuni campi importanti sono lo studio delle interazioni proteina-proteina, che dipendono principalmente dalle strutture terziarie e quaternarie delle proteine stesse e dalle interazioni dei loro domini. Inoltre, anche la purificazione delle proteine e l'analisi quantitativa dell'espressione proteica appartengono al campo della proteomica. Essa completa così i dati ottenuti nell'analisi dell'espressione genica e fornisce informazioni sui componenti delle vie metaboliche e dei sistemi di regolazione molecolare. L'ingegneria delle proteine permette di modificare le funzioni delle proteine ricombinanti per adattarne le proprietà.
Così, le tecniche principali della proteomica supportano lo studio della funzione e della struttura proteica 3-D e l'identificazione delle singole proteine.
Dato che tutti in processi metabolici le proteine giocano un ruolo fondamentale, gli approcci terapeutici contro il cancro, le infezioni e alcune malattie nervose si basano proprio su tali proteine. Malattie come l'anemia falciforme, il morbo di Alzheimer, la corea di Huntington o la malattia di Creutzfeldt-Jakob sono causate da un malfunzionamento di alcune proteine; quindi se sappiamo quale proteina è responsabile di una disfunzione, è possibile sviluppare specificamente una piccola molecola che si aggancia a questa proteina per prevenire le sue alterazioni. Le proteine ricombinanti sono anche usate nell'industria come enzimi detergenti e pesticidi biologici. I biologi sperano di ottenere una migliore comprensione del funzionamento degli organismi viventi e della vita in quanto tale. I biofisici si aspettano inoltre di sviluppare una "anatomia molecolare".
Biologia dei sistemi
Un nuovo campo di ricerca che si basa sulla proteomica è la biologia dei sistemi. Questa non guarda solo alle singole parti di una cellula, ma cerca di descrivere l'interazione di tutte le singole parti all'interno di un sistema e del suo ambiente. Oltre alla proteomica, questo approccio richiede modelli matematici che simulano il sistema in silico (cioè secondo modelli informatici), ma anche quelli che permettono di riprodurlo attraverso un approccio di tipo sperimentale.
Paleoproteomica
Le proteine fossili, oltre che il DNA stesso, possono occasionalmente essere isolate da ossa fossili, che permettono anche di trarre conclusioni, ad esempio, sulla loro appartenenza ad una particolare specie biologica. La paleoproteomica (dal greco παλαιός palaiós, "vecchio"), che si basa su questo, facendo leva sul fatto che alcune proteine sono più stabili del DNA per un periodo di tempo più lungo. Nel 2016, per esempio, il gruppo di ricerca di Jean-Jacques Hublin dell'Istituto Max Planck per l'antropologia evolutiva ha utilizzato campioni di collagene risalenti a circa 40.000 anni fa per chiarire che la cultura archeologica di Châtelperronia è legata ai Neanderthal e non all'uomo anatomicamente moderno (Homo sapiens). Nel 2019, le proteine fossili rilevate nella dentina della mandibola Xiahe, scoperta negli altipiani del Tibet nella grotta di Baishiya, hanno dimostrato che si trattava di esseri umani Denisova, e pochi mesi dopo, campioni di dentina di 1,9 milioni di anni hanno confermato che il genere Gigantopithecus è un taxon "sorella" estinto degli oranghi. Già nel 2015, le analisi del collagene hanno rivelato una relazione più stretta tra gli "ungulati sudamericani" e gli ungulati dalle dita dispari, cioè Macrauchenia e Toxodon, che erano ancora presenti nel tardo Pleistocene. In passato, le relazioni degli "ungulati sudamericani" con altri gruppi di ungulati erano risultate poco chiare e oggetto di dibattito scientifico. Per il rinoceronte estinto Stephanorhinus, i proteomi risalenti a circa 200.000 - 400.000 e 1,8 milioni di anni fa, rispettivamente nel 2017 e nel 2019 hanno rivelato una relazione più stretta con il rinoceronte lanoso e quindi un cerchio di parentela più stretto intorno al moderno rinoceronte di Sumatra. La posizione potrebbe essere supportata anche da studi genetici ed era stata precedentemente ipotizzata su basi anatomiche. Sempre nel 2019, gli studi sulle proteine hanno contribuito alla riclassificazione sistematica dei bradipi fossili e non.
Nel 2015, per esempio, uno studio sulle ossa di 80 milioni di anni fa di Brachylophosaurus canadensis, un membro del gruppo dei dinosauri dal becco d'anatra, ha attirato l'attenzione dei media di tutto il mondo. Lo studio ha rivelato che i loro peptidi , a causa della loro somiglianza con i peptidi trovati in uccelli e struzzi al giorno d‘oggi, sono stati interpretati come resti di vasi sanguigni.
Problemi e tendenze
Dopo alcune esperienze con metodi genetici come l'analisi microarray, alcuni scienziati si sono dimostrati scettici sulla ricerca sul proteoma. Friedrich Lottspeich del Max Planck Institute of Biochemistry di Martinsried, presidente della Società tedesca per la ricerca sul proteoma (DGPF), mette in guardia su false aspettative:
"Per il settore umano, la ricerca è in realtà ancora troppo complessa al momento comunque [...] Ma per un'analisi del lievito, che sarebbe un buon modello, ovviamente nessuno vuole spendere soldi".
La sua complessità deriva da quanto segue: secondo Friedrich Lottspeich, si stima che gli esseri umani abbiano diverse centinaia di migliaia o milioni di proteine diverse, un singolo gene esprime in media da cinque a dieci proteine, in alcuni casi diverse centinaia. Comprendere pienamente questa complessità è una sfida ancora troppo grande. D'altra parte, la ricerca sul proteoma si sta sviluppando rapidamente. Ciò è dovuto in particolare al costante miglioramento degli spettrometri di massa, che diventano sempre più precisi, sensibili e veloci.
Un altro passo importante è lo sviluppo di metodi quantitativi, come le procedure SILAC, iTRAQ, TMT o ICAT basate sull'uso di isotopi stabili o la codifica dei metalli MeCAT, in cui metalli di diverso peso sono usati per identificare proteine e peptidi da diversi campioni di proteine. Quest'ultimo permette l'uso del proteoma-wide della spettrometria di massa elementare ultrasensibile (ICP-MS) (limite di rilevamento nell'intervallo da ppt a ppq inferiore), che consente una maggiore sensibilità nella quantificazione delle proteine e ha un range di misura dinamica lineare che va da 6 a 8 ordini di grandezza. MeCAT, in contrasto con gli altri metodi, permette una assoluta quantificazione delle proteine. La calibrazione di ICP-MS viene eseguita utilizzando standard indipendenti dal rapporto proteina/peptide. La necessità di avere peptidi standard specifici per le proteine viene così eliminata.
Se l'analisi quantitativa del proteoma è combinata con altri metodi biologici, è anche possibile fare affermazioni sulla funzione delle proteine (ad esempio l'interazione proteina-proteina o le modifiche post-traslazionali). La moderna ricerca sul proteoma va quindi ben oltre la semplice catalogazione delle proteine e tenta di comprendere meccanismi complessi.
Applicazioni semplici
Per i ricercatori è importante poter quantificare le proteine. Una delle tecniche più importanti per questo è l'elettroforesi su gel di poliacrilammide (PAGE), con questa tecnica le proteine sono separate in base alle loro dimensioni in un campo elettrico. Siamo in grado di offrire tutto il necessario per preparare del gel per elettroforesi. Troverete una selezione di soluzioni di acrilammide e bisacrilammide, APS, TEMED e materiali di supporto DTT, β-mercaptoetanolo, Coomassie oltre ai marcatori di di proteine.
Le proteine sono sempre predeterminate nella loro modalità d'azione e disponibilità funzionale, pertanto, le proteine devono prima di tutto essere isolate dal resto per poter essere studiate, questo può avvenire da un lato con metodi fisici, come la rottura delle cellule con il calore o chimicamente. Per esempio, il nostro prodotto Tritidy G può aiutare a isolare simultaneamente DNA, RNA e proteine da un campione biologico. Tuttavia, i detergenti sono particolarmente importanti in questo contesto. Sono disponibili molti detergenti ben noti come SDS e Tween, ma anche altri più specifici come Digitonin e Saponin, che sono spesso utilizzati per le proteine di membrana. Vorremmo anche segnalare i nostri prodotti sostitutivi del Triton X-100.
Prodotti alternativi al Triton X-100:
| CODICE DEL PRODOTTO | NOME DI PRODOTTO | NUMERO CAS |
| A9778 | ECOSURF™ EH-9 (sostituzione del Triton X-100) |
64366-70-7 |
| A9779 | ECOSURF™ SA-9 (sostituzione del Triton X-100) |
|
| A9780 | TERGITOL™ 15-S-9 (sostituzione del Triton X-100) |
68131-40-8 |
L'originale, Triton X-100 è stato classificato come una sostanza ad alto rischio e sotto regolamento REACH e non può più essere venduto facilmente.
Le tecniche di biochimica delle proteine non sono solo utilizzate per determinare gli stati naturali delle proteine, ma anche per produrre proteine desiderate per mezzo di manipolazioni mirate. A questo scopo vengono utilizzati i cosiddetti sistemi di espressione. Uno dei più noti è il Lac-operon: i geni per certe proteine sono assemblati dalla biologia molecolare utilizzando questo operone. Nei batteri (E. coli), l'organismo produce poi le proteine. L'intero processo è indotto dalla sostanza IPTG (isopropyl-β-D-thiogalactopyranoside). Abbiamo una selezione di IPTG disponibile per i nostri clienti.
| CODICE DEL PRODOTTO | NOME DI PRODOTTO | NUMERO CAS |
| A1008 | IPTG BioChemica | 367-93-1 |
| A4773 | IPTG per biologia molecolare, senza diossano | 367-93-1 |
| A7211 | IPTG da galattosio di origine vegetale, senza diossano | 367-93-1 |
Il più grande nemico di una proteina è ... un'altra proteina. La degradazione delle proteine è infatti catalizzata dalle proteasi. Queste si trovano in tutti gli organismi e quindi spesso anche nei campioni stessi, per tale motivo vengono utilizzati i cosiddetti inibitori. Questi interferiscono con l'attività catalitica delle proteasi in modo che queste non possano diventare attive.
A volte, tuttavia, è proprio la degradazione delle proteine che si desidera ottenere, ad esempio, se si vogliono esaminare solo gli acidi nucleici in un campione, le proteine possono interferire. Un esempio ben noto potrebbe essere rappresentato da alcuni kit di test per la ricerca COVID-19. La proteinasi K è una proteasi che catalizza la degradazione delle proteine. Abbiamo a disposizione una Proteinase K di alta qualità in forma liofilizzata e una già disciolta pronta all'uso.
Altri prodotti importanti che vengono utilizzati sono le albumine e i cofattori come il NAD e il NADPH, questi possono essere necessari come stabilizzatori o come coenzimi in varie reazioni.
Il campo della proteomica rimarrà un focus per molto tempo, ci mostra il vero stato reale di ogni cellula. Molti saggi porteranno ancora a scoperte rivoluzionarie.
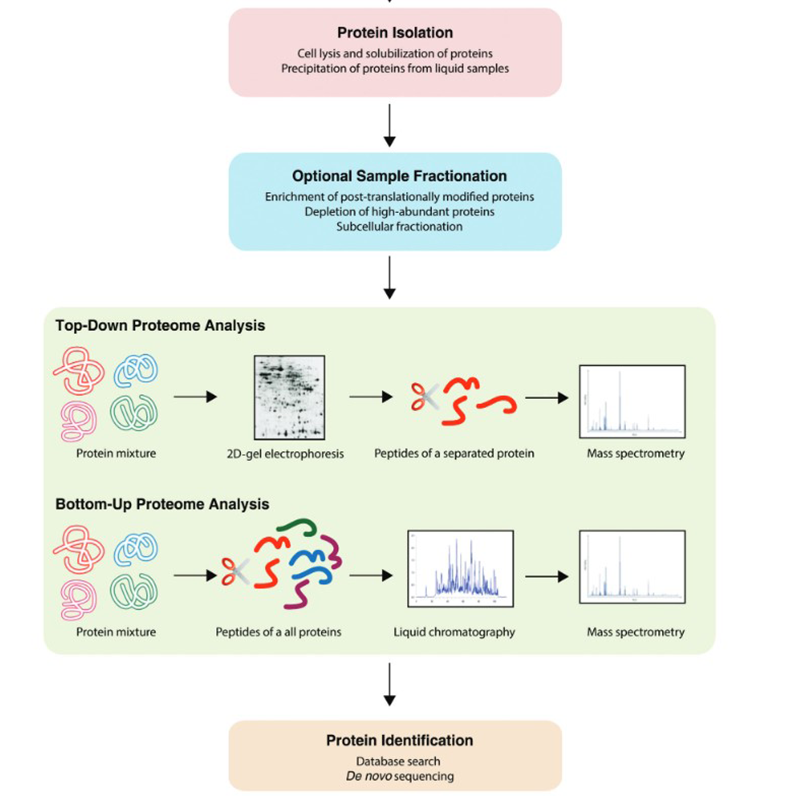
Oltre alle tecniche già menzionate sopra, una varietà di applicazioni può essere trovata nel campo della proteomica. Queste possono avvenire dal basso verso l'alto o dall'alto verso il basso:
.png)
La proteomica viene applicata sia su cellule che su cellule che animali. L'elettroforesi su gel 2D viene molto utilizzata. Grandi passi nel campo della proteomica sono stati fatti con metodi cromatografici accoppiati alla spettrometria di massa. A questo scopo offriamo i solventi più comuni.
I metodi comunemente usati nella proteomica oggi includono la cromatografia, la spettrometria di massa, l'imaging MALDI, l'elettroforesi di poliacrilammide unidimensionale (1D PAGE), l'elettroforesi di gel di poliacrilammide bidimensionale (2D PAGE), i microarray, l'analisi degli aminoacidi (AAA) e molto altro.
La proteomica qualitativa e quantitativa vieneutilizzata nella disgnosi di tumori e permette di trarre conclusioni per prognosi e terapie per i singoli pazienti.
Le proteine sono anche di grande importanza in immunologia basti pensare che i nostri anticorpi sono forme speciali di proteine.
Secondo Friedrich Lottspeich (biochimico austriaco), si stima che gli esseri umani abbiano diverse centinaia di migliaia o milioni di proteine diverse. Un singolo gene esprime in media da cinque a dieci proteine, in alcuni casi diverse centinaia. Cogliere appieno questa complessità è una sfida che i metodi attuali non sono ancora all'altezza. D'altra parte, la ricerca sul proteoma si sta sviluppando rapidamente. Ciò è dovuto in particolare al costante miglioramento degli spettrometri di massa, che diventano sempre più precisi, sensibili e veloci.
Una panoramica dei prodotti adatti alla ricerca sulle proteine (proteomica) può essere trovata qui sotto:
| CODICE DEL PRODOTTO | NOME DI PRODOTTO | NUMERO CAS |
| A1272 | 2-Nitrofenil-β-D-Galattopiranoside BioChemica | 369-07-3 |
| A3840 | 3,3',5,5'-Tetrametilbenzidina BioChemica | 54827-17-7 |
| A1523 | 4-Aminoantipirina BioChemica | 83-07-8 |
| A1028 | 4-Nitrofenil-β-D-Glucuronide BioChemica | 10344-94-2 |
| A1088 | ABTS® BioChemica | 30931-67-0 |
| A4983 | Acrilammide soluzione (30%) - Miscela 29 : 1 per biologia molecolare | |
| A3626 | Acrilammide soluzione (30%) - Miscela 37,5 : 1 per biologia molecolare | |
| A3658 | Acrilammide soluzione (40%) - Miscela 19 : 1 per biologia molecolare | |
| A0385 | Acrilammide soluzione (40%) - Miscela 29 : 1 per biologia molecolare | |
| A4989 | Acrilammide soluzione (40%) - Miscela 37,5 : 1 per biologia molecolare | |
| A7582 | Acrilammide 2K soluzione (18%) per denaturare il DNA-PAGE | |
| A7590 | Acrilammide soluzione 2K (8%) per denaturare il DNA-PAGE | |
| A1089 | Acrilammide 2K Grado standard, extrapuro | 79-06-1 |
| A0951 | Acrilammide 4K soluzione (30%) - Miscela 29 : 1 | |
| A1672 | Acrilammide 4K soluzione (30%) - Miscela 37,5:1 | |
| A1577 | Acrilammide 4K soluzione (40%) - Miscela 37,5:1 | |
| A1090 | Acrilammide 4K ultrapuro | 79-06-1 |
| A1421 | AEBSF cloridrato BioChemica | 30827-99-7 |
| A0850 | Albumina (BSA) grado EIA e RIA | 9048-46-8 |
| A2244 | Albumina (BSA) Frazione V (pH 5.2) | 9048-46-8 |
| A1391 | Albumina (BSA) Frazione V (pH 7.0) | 9048-46-8 |
| A6588 | Albumina (BSA) Frazione V (pH 7.0) per Western blotting | 9048-46-8 |
| A4344 | Albumina grezza da albume d'uovo di gallina | 9006-59-1 |
| A7708 | AppliCoat Plate Stabilizer | |
| A2132 | Aprotinina BioChemica | 9087-70-1 |
| A2568 | Avidina BioChemica | 1405-69-2 |
| A1117 | BCIP BioChemica | 6578-06-9 |
| A3636 | Bisacrilammide per biologia molecolare | 110-26-9 |
| A3417 | CheLuminate-HRP PicoDetect | |
| A2144 | Chymostatin | 9076-44-2 |
| A1101 | DTT BioChemica | 3483-12-3 |
| A2948 | DTT per biologia molecolare | 3483-12-3 |
| A3668 | DTT soluzione 1 mol/L (1 M) per biologia molecolare | 3483-12-3 |
| A1008 | IPTG BioChemica | 367-93-1 |
| A4773 | IPTG per biologia molecolare, senza diossano | 367-93-1 |
| A7211 | IPTG da galattosio di origine vegetale, senza diossano | 367-93-1 |
| A2243 | L-Glutatione ossidato BioChemica | 27025-41-8 |
| A9782 | L-Glutatione ridotto (USP) puro, grado farmaceutico | 70-18-8 |
| A2084 | L-Glutatione ridotto BioChemica | 70-18-8 |
| A2185 | Luminol | 521-31-3 |
| A1243 | NBT BioChemica | 298-83-9 |
| A0830 | Latte secco magro in polvere | |
| A2205 | Pepstatin A | 26305-03-3 |
| A0999 | PMSF BioChemica | 329-98-6 |
| A2935 | Ponceau S - Soluzione | |
| A8889 | Marcatore della proteina VI (10 - 245) pre-colorato | |
| A1108 | β-Mercaptoetanolo per biologia molecolare | 60-24-2 |
| A1495 | Streptavidina ultrapura | 9013-20-1 |
| A1148 | TEMED | 110-18-9 |
| A1828 | Inibitore della tripsina della soia > 7000 BAEE | 9035-81-8 |
| A1007 | X-Gal BioChemica | 7240-90-6 |
| A4978 | X-Gal per biologia molecolare | 7240-90-6 |