ITW Reagents präsentiert die DNA-ExitusPlusTM Technology
DNA-ExitusPlusTM & DNA-ExitusPlusTM IF
Reagenz zur Entfernung von DNA- und RNA-VerunreinigungenProduktnummern A7089 und A7409
Verfügbare Literatur:
InfoPoint
Produkt-Info
Broschüre
Laborposter (bestellen Sie Ihr Exemplar bei Ihrem örtlichen Händler)
Beschreibung
DNA-ExitusPlus™ ist ein patentiertes Reagenz zur Entfernung von DNA- und RNA-Verunreinigungen von Laboroberflächen und -geräten (1). Die Lösung verwendet eine milde und nicht korrosive Chemie für einen schnellen, nicht-enzymatischen Abbau von Nukleinsäuren. Bereits kurze Inkubationszeiten mit DNA-ExitusPlus™ entfernen unerwünschte DNA und RNA vollständig von Arbeitsflächen und Werkzeugen (1, 2).Es gibt zwei verschiedene Versionen von DNA-ExitusPlus™: DNA-ExitusPlus™ (A7089) enthält einen Farbindikator, um die mit dem Reagenz bedeckte Oberfläche leicht sichtbar zu machen. DNA-ExitusPlus IF™ (A7409) ist fast farblos. Beide Lösungen verdunkeln sich mit der Zeit aufgrund der in den Lösungen enthaltenen redoxaktiven Komponenten.
Hinweis: Es gibt keine Unterschiede in den Anwendungsprotokollen von DNA-ExitusPlus™ und DNA-ExitusPlus IF™! Daher nennen wir die IF-Bezeichnung in der folgenden Gebrauchsanweisung nicht.
Die einzigartigen Eigenschaften von DNA-ExitusPlus™

- Katalytische und kooperative Effekte der Komponenten führen zu einem sehr schnellen, nicht-enzymatischen, sequenzunabhängigen Abbau von DNA- und RNA-Molekülen, die alle Verunreinigungen entfernen.
- Alle Komponenten von DNA-ExitusPlus™ sind leicht biologisch abbaubar und für den Menschen nicht schädlich oder toxisch.
- Es werden keine aggressiven Mineralsäuren oder alkalischen Substanzen verwendet. Geräte und Materialien werden auch nach längerer Inkubationszeit nicht beschädigt oder korrodiert.
- Keine giftigen Dämpfe.
- Erhöhte Temperaturen über ca. 50°C beschleunigen die Reaktion und die Aktivität.
Anwendungshinweise
- Die optimale Inkubationszeit für die Dekontamination von Oberflächen beträgt 10 Minuten und eine Temperatur über 20°C. Nach der Inkubation wird der Rest DNA-ExitusPlusTM mit einem Papiertuch abgewischt. Eine zusätzliche Reinigung mit sterilem Wasser ist danach nicht mehr erforderlich. Dies ist eine Neuerung gegenüber den herkömmlichen Dekontaminationslösungen.
- Nach vollständiger Trocknung der Lösung findet keine weitere Dekontaminationsreaktion statt. Eine Inkubationszeit von mehr als 30 Minuten ist daher nicht notwendig und auch nicht sinnvoll. Bei starken Verunreinigungen wird eine zweite Anwendung der Lösung für höchste Effizienz empfohlen.
- Zur Entfernung unerwünschter, getrockneter Restspuren des Reagenzes empfehlen wir, diese Spuren mit sterilem Wasser oder 10X TE-Puffer und einem Papiertuch zu entfernen.
Zur Dekontamination von Laborflächen: Tragen Sie DNA-ExitusPlus™ direkt auf die Laboroberfläche auf. Inkubation für 10 Minuten. Restliches DNA-ExitusPlus™ mit einem Papiertuch gründlich abwischen (getrocknete Rückstände mit sterilem Wasser / 10X TE-Puffer entfernen). Eine Spülung mit Wasser ist nicht erforderlich.
Zur Dekontamination von Laborgeräten: Tragen Sie DNA-ExitusPlusTM großzügig auf ein Papiertuch auf und wischen Sie alle exponierten Oberflächen des Gerätes gründlich ab. Mit einem sauberen Papiertuch trocknen. Um kleine Teile zu reinigen, tauchen Sie sie kurz in DNA-ExitusPlusTM ein und trocknen Sie sie.
Zur Dekontamination von Kunststoff- und Glasbehältern: Fügen Sie reichlich DNA-ExitusPlusTM hinzu, um die gesamte Oberfläche des Gefäßes durch Verwirbeln zu beschichten. Entsorgen Sie die Lösung und lassen Sie die Gefäße trocknen. Spülen Sie die Behälter gründlich mit destilliertem Wasser ab und trocknen Sie sie.
Zur Dekontamination von Pipetten: Befolgen Sie die Anweisungen des Herstellers, entfernen Sie den Schaft von der Pipette und entfernen Sie Dichtungen und Manschetten von der Welle. Den Schaft eine Minute lang in DNA-ExitusPlusTM einweichen, dann gründlich mit Wasser abspülen, trocknen lassen und wieder zusammenbauen.
Eine neue Technologie zur Dekontamination von Nukleinsäure
Die DNA-Amplifikation ist eine der am häufigsten verwendeten Techniken im modernen Forschungslabor. Das Vorhandensein von kontaminierender DNA in und um PCR-Arbeitsplätze herum kann zu unerwünschten Artefakten während der Amplifikation führen. Grundsätzlich gibt es zwei Möglichkeiten, die DNA nicht amplifizierbar zu machen:- durch Abbau der DNA (z. B. durch Zugabe von DNasen oder chemische Zerstörung) oder
- durch Modifikation der Basen - wobei der DNA-Strang intakt bleibt, aber zum Lesen durch Polymerasen blockiert ist.
Mit einem DNA-Strangbruch-Assay (entwickelt von der multiBIND biotec GmbH, Deutschland; siehe Abb. 1) wurde gezeigt, dass nicht alle auf dem Markt befindlichen DNA-Dekontaminationslösungen die DNA vollständig abbauen! DNA-ExitusPlus™ ist eine Verbesserung gegenüber diesen Produkten und verursacht sowohl Strangbrüche als auch Degradationen. Bei richtiger Anwendung im Arbeitsbereich entfällt die Amplifikation von Nicht-Ziel-DNA vollständig (Abb. 3). DNA-ExitusPlusTM ist eine alkalifreie, nicht korrosive und nicht krebserregende Reinigungslösung, die auf allen Oberflächen wirksam ist.
Ein gravierender Nachteil herkömmlicher Dekontaminationsreagenzien zeigt sich in einem neuen Test auf das korrosive Potenzial ihrer Komponenten. Zu diesem Zweck wurden verschiedene Metallplatten für 20 Minuten mit identischen Aliquoten der Reagenzien inkubiert. Die ausgewählten Metalle sind repräsentativ für Geräte und Materialien, die in Labors verwendet werden. Das Ergebnis dieser Korrosionsprüfung ist in Abbildung 2 dokumentiert und zeigt, dass alle bekannten handelsüblichen Produkte aggressive chemische Substanzen mit korrosiven, schädlichen oder sogar toxischen Auswirkungen enthalten. Diese herkömmlichen Reagenzien enthalten bekanntlich Azide, Mineralsäuren wie Phosphorsäure oder Salzsäure, aggressive Peroxide oder stark alkalische Substanzen wie Natriumhydroxid. Bereits nach einer Inkubation von nur 20 Minuten sind in vielen Fällen irreversible Schäden an den Metalloberflächen zu beobachten (siehe Abbildung 2). Die neu entwickelte Lösung DNA-ExitusPlusTM zeigt insbesondere bei diesem Korrosionstest ihre einzigartigen Eigenschaften. Bei allen getesteten Metalloberflächen ist nach der Behandlung keine Beschädigung oder Korrosion zu beobachten. DNA-ExitusPlusTM wurde auch unter identischen Bedingungen auf vielen verschiedenen Kunststoffoberflächen ohne Anzeichen von Schäden getestet (Daten nicht dargestellt).
Das Reagenz ist mindestens 12 Monate haltbar und hitzebeständig.
(1) Esser, K.-H. et al. (2006) DNA decontamination: DNA-ExitusPlus™ in comparison with conventional reagents. Nature Methods 3, 151
(2) Arena, A. (2010) DNA-ExitusPlus™ versus standard bleach solution for the removal of DNA contaminants on work surfaces and tools. Investigative sciences journal 2, 20-29
Qualitätsprüfung
Quantifizierung des DNA-Abbaus durch analytische Agarosegelelektrophorese oder PCREs wird immer wichtiger, den DNA-Abbau nach Dekontaminationsmaßnahmen im Labor genau zu kontrollieren.
Wird DNA mit DNA-ExitusPlus™ oder einem anderen Dekontaminationsreagenz inkubiert und eine Probe des Reaktionsgemischs ohne vorherige Neutralisation oder Denaturierung auf ein Agarosegel aufgetragen, ist eine quantitative Bestimmung des DNA-Abbaus nicht möglich. Viele Fragmente werden auch nach Strangbrüchen zu größeren Einheiten zusammenkleben (10 bis 20 homologe Basenpaare reichen aus, um durch Bildung größerer Einheiten zu hybridisieren). Dieses Phänomen der Hybridisierung wird bei DNA-Fragmenten mit klebrigen Enden beobachtet, z.B. des λ DNA-Größenmarkers für die Gelelektrophorese. Aus diesem Grund wird eine solche Marker-DNA vor dem Auftragen auf ein Gel denaturiert.
Die Reagenzien anderer Anbieter enthalten häufig hohe Konzentrationen an starken Säuren oder Basen. Wenn Sie diese Reaktionsgemische vor der Verabreichung an das Gel nicht neutralisieren, beobachten Sie eine Farbänderung des pH-Indikators Bromphenolblau (Übergangsintervall bei pH 2 - 4,6 von grünlich-gelb zu blau-violett). Wenn Sie eine nicht neutralisierte Probe auf ein Gel geben, können Sie die Zerstörung des Schlitzes durch die Chemikalien beobachten. Ethidiumbromid wird unter diesen Bedingungen zerstört und kann daher nicht angewendet werden, da es keine DNA-Färbung verursacht. Die Bahnen auf dem Gel wären klar, auch wenn große Mengen an ungefärbter DNA vorhanden sind. Durch die Neutralisierung der Probe mit Tris-Puffer wird die richtige Farbe von Bromphenolblau sichtbar. Je nach Zusammensetzung des Dekontaminationsreagenzes muss die Neutralisation entweder mit 100 mM Tris pH 12 oder 100 mM Tris pH 3 durchgeführt werden. Im Falle von DNA-ExitusPlus™ reicht die Pufferkapazität des Ladepuffers aus, um selbst eine 1:1-Mischung aus Dekontaminationsreagenz und Probe zu puffern.
Wenn die Neutralisation von Bromphenolblau den richtigen pH-Wert zeigt, werden die Proben für 2 Minuten bei 90°C denaturiert.
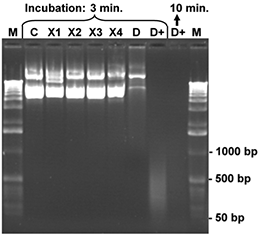
Aliquots einer CCC-Plasmid-DNA (7 kb), 200 ng für jede Probe, wurden in 10 µl Wasser gelöst und mit 5 µl eines der aufgeführten Reagenzien für 3 bzw. Minuten bei Raumtemperatur behandelt.Schließlich wurden die Proben mit Bromphenolblau-Ladepuffer gemischt und für 3 Minuten bei 92°C denaturiert. Die denaturierten Proben wurden auf Eis gelegt und das komplette Reaktionsgemisch auf ein 1 %iges Agarosegel aufgetragen. Nach der Gelelektrophorese wurde das Agarosegel mit Ethidiumbromid gefärbt und fotografiert. Die Kontrolle (C) enthält die intakte CCC-Plasmid-DNA (200 ng) nach der Behandlung mit 5 µl sterilem Wasser. Schnitte und Beschädigungen der DNA-Stränge erzeugen Fragmente mit geringerem Molekulargewicht. Diese kleineren DNA-Fragmente können im Gel durch Vergleich mit der Kontrollprobe und dem Molekulargewichtsmarker (M; 1 kb Leiter) identifiziert werden. Die Produkte X1-X4 verursachen nur einen sehr geringen Abbau der Test DNA, während Produkt D (konventionelles DNA-Exitus) unter diesen Bedingungen effektiver war. Nur DNA-ExitusPlusTM (D+) bewirkt einen sehr schnellen und nahezu vollständigen DNA-Abbau nach 3 Minuten. Dabei werden nur Rest-DNA-Fragmente kleiner als 500 Basenpaare beobachtet. Eine verlängerte Inkubation (10 Minuten) zerstörte die gesamte Plasmid-DNA.
Die hohe Konzentration von Chemikalien in den Reagenzien anderer Anbieter führt zu schwerwiegenden Problemen bei der PCR-Analyse. Proben, die durch Abwischen von mit solchen Reagenzien behandelten Laborbänken gesammelt wurden, enthielten Chemikalien in so hohen Konzentrationen, dass die PCR auch nach der Verdünnung gehemmt wurde. Daher ist eine positive Kontrolle der PCR mit einer Mischung aus einem definierten Template und einem Aliquot der gesammelten Probe erforderlich, um falsch negative Ergebnisse auszuschließen. In unseren Händen enthielt eine solche Negativkontroll-PCR noch Bestandteile der Reagenzien in hemmenden Konzentrationen, was eine weitere Verdünnung oder Neutralisation erforderlich machte.
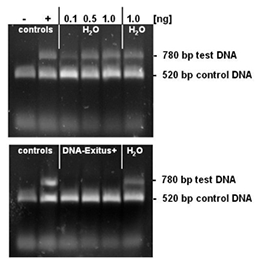
Ausgewählte Mengen (0,1 bis 1 ng) einer Test DNA wurden auf der Innenfläche von PCR-Röhrchen lyophilisiert. Die PCR-Gefäße mit der lyophilisierten DNA-Probe wurden 20 Sekunden lang entweder mit sterilem Wasser oder DNA-ExitusPlusTM inkubiert. Danach wurden die Röhrchen zweimal mit 100 µl sterilem Wasser gewaschen. Für die PCR-Testreaktion wurden Mischungen von 50 µl in jedes Röhrchen gegeben. Dieses Reaktionsgemisch enthält Primer zur Amplifikation der Kontroll-DNA (Kontrolle) und der Test-DNA. Die Kontroll-DNA (1 ng) wird jeder Probe beigefügt und beweist, dass die PCR-Reaktion nicht gehemmt ist. Die Amplifikation einer DNA-Bande, die der Test-DNA entspricht, zeigt an, dass intakte DNA-Moleküle dieser Vorlage noch vorhanden sind. Nach vollständigem Abbau und Entfernung der Test-DNA sollte die PCR-Reaktion kein DNA-Fragment für diese Vorlage amplifizieren. Nach der Elektrophorese durch ein 1%iges Agarosegel wurde das Gel mit Ethidiumbromid gefärbt und dokumentiert. Die Negativkontrolle mit sterilem Wasser (H2O) zeigt DNA-Bänder für die Test- und Kontrollvorlagen. Die PCR-Reaktion nach der Behandlung mit DNA-ExitusPlus™ amplifiziert nur das Fragment der Kontroll-DNA. Dies zeigt, dass die Behandlung mit DNA-ExitusPlus™ alle Spuren der Test DNA-Vorlage zerstört und entfernt..

Für diese Prüfung wurden Metallplatten ausgewählt, die typisch für Labormaterialien und -geräte sind. Aliquots von 10 µl aus jedem aufgeführten Reagenz wurden auf die verschiedenen Metalloberflächen aufgebracht. Als Kontrolle wurde steriles Wasser verwendet (0). Nach einer Inkubationszeit von 20 Minuten wurden die Reagenzien abgewischt und die Metalle kurz mit sterilem Wasser gewaschen. Nach vollständiger Trocknung wurden die Metallplatten fotografiert. Die Reagenzien X2, X3 und D (siehe Abbildung 2) zur DNA-Dekontamination verursachen irreversible Korrosion und Schäden an vielen Metalloberflächen. Unter DNA-ExitusPlus™ (D+) ist keine Beschädigung einer der Oberflächen zu beobachten. In einigen Fällen beobachtet man einen Poliereffekt durch die Entfernung von Schmutz- oder Oxidschichten.
Abbau von kleinen DNA-Fragmenten
Die Strangbruchaktivität von DNA-ExitusPlusTM ist unabhängig von der Größe der DNA-Fragmente, da die Zerstörung auf einer chemischen und nicht auf einer enzymatischen Aktivität basiert. Daher wurde ein 750 bp PCR-Produkt für unterschiedliche Zeiten mit DNA-ExitusPlusTM inkubiert (siehe Abb. 4). Wie erwartet, können die Primer auch nach kürzester Inkubationszeit nicht erkannt werden. Nach 5 Minuten Inkubation mit DNA-ExitusPlusTM wird fast die gesamte DNA zerstört. Wir wollen deutlich machen, warum das so ist: Unter der Annahme, dass eine theoretische Schneide-Aktivität von 100.000 Schnitten pro Minute vorliegt, werden alle DNA-Fragmente zerstört, unabhängig von ihrer Größe. Kleinere Fragmente werden zuerst verschwinden, dann die größeren. Übertragen auf ein Testmolekül (ccc-Form, 6 kb Plasmid) bleibt daher nach 5 Minuten ein kleiner Teil der Fragmente mit nur 200 bis 500 bp Größe übrig. Die Schnitte werden statistisch an jeder Stelle eingeführt, so dass keine einzige Klasse von Fragmenten übrig bleibt. Daher ist die PCR-Analyse negativ. Beim Sprühen von DNA-ExitusPlusTM auf Oberflächen im Labor wird ein enormer Überschuss von ca. 1 bis 5 ml Reagenz auf kleinste DNA-Mengen aufgetragen.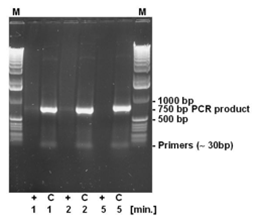
Zur Untersuchung des Abbaus kleiner DNA-Fragmente wurden 500 ng DNA einer PCR-Reaktion, die ein 750 bp PCR-Fragment ergibt, für die angegebenen Zeiträume (1, 2 und 5 Minuten) mit DNA-ExitusPlusTM inkubiert. Nach der Behandlung wurde die DNA für 2 Minuten bei 95°C denaturiert. (+) 5 µl DNA plus 5 µl DNA-ExitusPlusTM; (C) Kontrolle 5 µl DNA plus 5 µl Wasser; (M) Molekulargewichtsmarker 1 kb Leiter.
Abbau von RNA durch DNA-ExitusPlus™
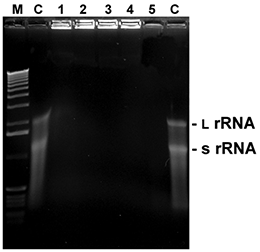
Abb. 5. RNA Abbau
5 µl mit 1 µg Gesamt-RNA von E. coli wurden mit 5 µl der aufgeführten Reagenzien gemischt und bei RT für die angegebene Zeit inkubiert. Danach wurden die Proben mit Ladepuffer gemischt, für 2 Minuten auf 60°C erhitzt und sofort auf ein 1,5%iges Agarosegel mit Formamid/Formaldehyd als Denaturierungsmittel aufgetragen. Nach dem Waschen und Renaturieren der Gel-RNA wurde diese durch EtBr-Färbung visualisiert (L rRNA: große ribosomale RNA; s rRNA: kleine ribosomale RNA).
M: 1 kb Leiter
C: Kontrolle (steriles Wasser)
1:D+ 0.5 Minuten
2:D+ 1 Minute
3:D+ 2 Minuten
4:D+ 5 Minuten
5: RNaseA (10 ng / 5 Minuten)
Effizienzsteigerung durch erhöhte Inkubationstemperatur
Es ist äußerst schwierig, DNA zu entfernen und zu zerstören, wenn sie auf einer Oberfläche getrocknet ist oder DNA, die durch z.B. die Hülle eines Virus "geschützt" ist. Selbst das Autoklavieren kann diese DNA in manchen Fällen nicht abbauen. Das komplette Genom eines Vogelvirus konnte nach dem Autoklavieren nachgewiesen werden [Elhafi, G., Naylor, C.J., Savage, C.E. und Jones, R.C. (2004). Mikrowellen- oder Autoklavbehandlungen zerstören die Infektiosität des infektiösen Bronchitisvirus und des Vogelpneumovirus, ermöglichen aber den Nachweis durch Reverse-Transkriptase-Polymerase-Kettenreaktion. Vogelpathologie 33, 303-306]. Die Aktivität von DNA-ExitusPlus™ als Zusatz zu Lösungen beim Autoklavieren wurde getestet. Es konnte gezeigt werden, dass erhöhte Temperaturen die DNA-Abbaubarkeit von DNA-ExitusPlus™ verbessert haben!
50 ml Kulturen von rekombinanten E. coli-Stämmen wurden bei 120°C und 1,2 bar für 20 Minuten unter Zugabe gleicher Mengen an Wasser (-) oder DNA-ExitusPlusTM (+) autoklaviert. Anschließend wurden 10 µl Aliquots dieser Kulturen mit analytischen DNA-Agarosegelen untersucht. Die Probe mit sterilem Wasserzusatz (-) zeigt große Mengen an hochmolekularen DNA-Fragmenten. Die Zugabe des gleichen Volumens an DNA-ExitusPlusTM (+) führt zum Abbau der DNA und zeigt Fragmentgrößen unter 20 Basenpaaren.

Die autoklavierten rekombinanten E. coli-Kulturen aus Abb. 5 enthalten ein Plasmid mit dem Ampicillin-Resistenzgen (AmpR-Gen). Aliquots (2 µl) der Kulturen wurden in einer PCR-Reaktion mit Primern für das gesamte AmpR-Gen nach dem Autoklavieren getestet. Die Probe mit sterilem Wasser (-) ergibt eine starke Bande des gesamten AmpR-Gens (930 bp), während mit DNA-ExitusPlusTM (+) behandelte Proben ein positives Ergebnis verhinderten. Als Positivkontrolle (K) wurde ein 2 µl Aliquot der Probe mit DNA-ExitusPlusTM mit 2 ng Template DNA gemischt, die für das AmpR-Gen kodiert. Die Amplifikation der entsprechenden Bande zeigt, dass die PCR-Reaktion unter den Testbedingungen funktioniert.
FAQs
Wie ist die vorgeschlagene Kontaktzeit?Für Verunreinigungen mit geringen Mengen an DNA sind 5 bis 10 Minuten bei Raumtemperatur ausreichend. Bei größeren Mengen an DNA wird empfohlen, den Vorgang nach dem Abwischen von DNA-ExitusPlusTM zu wiederholen.
Darüber hinaus erhöht die Erhöhung der Temperatur der Lösung auf 50°C oder 60°C die Abbaurate der DNA erheblich.
Gibt es irgendwelche Methoden zum Nachweis von Resten der Komponentenmaterialien von DNA-ExitusPlusTM?
Die neue Rezeptur von DNA-ExitusPlusTM (Stand Oktober 2009) enthält eine geringe Menge an Farbindikator, der auf Oberflächen nach dem Trocknen nachgewiesen werden kann. Müssen kleine Restspuren von DNA-ExitusPlusTM entfernt werden, können diese leicht mit Filterpapier, das in sterilem Wasser getränkt ist, von den Oberflächen abgewischt werden.
Zur Inaktivierung größerer Restmengen von DNA-ExitusPlusTM können die Oberflächen mit einer Lösung aus sterilem 10X TE-Puffer, pH 8,0, behandelt werden. Achten Sie darauf, dass bei dieser Behandlung neue Verunreinigungen vermieden werden.
Ihre Ausrüstung umfasst Dichtungen aus Polypropylen, Edelstahl, EPDM und Silikon. Welche Informationen haben wir über die "Beschichtung" oder das Anhaften an diesen Materialien?
EPDM = Ethylen-Propylen-Dien-Kautschuk
Polypropylen-, Edelstahl-, EPDM- und Silikondichtungen werden durch DNA-ExitusPlusTM nicht beschädigt und die Lösung kann leicht mit sterilem Wasser oder 10X TE-Puffer, pH 8,0, abgewaschen werden. Auf diesen Oberflächen sind Restmengen von DNA-ExitusPlusTM getrocknet, die in einem einzigen Waschschritt mit den gleichen Lösungen entfernt werden können. Die Lösung kann dann mit sauberen Papiertüchern abgewischt werden.
Dünne Röhrchen - wie kann ich überschüssiges DNA-ExitusPlusTM entfernen?
DNA-ExitusPlusTM hat vor allem bei Röhrchen mit kleinem Durchmesser den Vorteil gegenüber anderen kommerziellen Produkten, dass die Ionenstärke aller Komponenten wesentlich geringer ist als bei anderen kommerziellen Produkten.
Darüber hinaus sind alle Komponenten von DNA-ExitusPlus™ hochgradig wasserlöslich und verändern die Viskosität von Wasser nicht wesentlich. Sie alle haben eine sehr geringe Affinität zu Metall- oder Kunststoffoberflächen.
Daher reicht auch für Röhrchen mit sehr kleinem Durchmesser ein Spülschritt mit 10X TE-Puffer, pH 8,0, und eine abschließende Spülung mit sterilem Wasser für die vollständige Entfernung von DNA-ExitusPlus™ .
Als letzte Kontrolle bestimmen Sie den pH-Wert des sterilen Wassers nach der letzten Spülung. Ein pH-Wert zwischen 6 und 8 ist akzeptabel. Bitte überprüfen Sie vor der Anwendung den pH-Wert des sterilen Wassers. Achtung: Steriles Wasser, das mit Anionenaustauschharzen hergestellt wird, kann einen niedrigen pH-Wert aufweisen.
Warum sieht man eine helle Fluoreszenz in den Schlitzen der mit DNA-ExitusPlusTM behandelten Proben aus Abb. 5?
Die in den Schlitzen des Agarose-Gels beobachtete Fluoreszenz wird durch Bestandteile des restlichen DNA-ExitusPlusTM, nicht aber durch DNA oder RNA verursacht. Diese kleinen Moleküle diffundieren trotz Elektrophorese in alle Richtungen in die Gelmatrix. Im Gegensatz dazu würde sich die DNA am Boden des Schlitzes ansammeln, falls sie ausfällt. Unter den gewählten Versuchsbedingungen würden sich weder RNA noch DNA ausfällen, und die RNA-Proben dieses Experiments enthalten keine hochmolekulare DNA, die in den Schlitzen zurückgehalten wird.